Zytosinstruktur, Funktionen, Eigenschaften, Synthese
- 2892
- 605
- Jasmine Kupprion
Der Zytosin Es handelt. Diese Verbindungen dienen jeweils Biosynthese aus Desoxyribonukleinsäure (DNA) und Ribonukleinsäure (RNA). DNA speichert genetische Informationen und RNA hat verschiedene Funktionen.
In Lebewesen wird Cytosin nicht frei gefunden, sondern häufig Ribonukleotide oder Desoxyribonukleotide. Beide Arten von Verbindung haben eine Phosphatgruppe, eine Ribose und eine Stickstoffbase.
 Quelle: Vesprcom [Public Domain]
Quelle: Vesprcom [Public Domain] Der Kohlenstoff 2 der Ribose hat eine Oxhydrillo (-OH) -Gruppe in den Ribonukleotiden und ein Wasserstoffatom (-H) in den Desoxyribonukleotiden. Abhängig von der Anzahl der vorhandenen Phosphatgruppen gibt es Citidín-5'-Monophosphat (CMP), Citidín-5'-Diphosphat (CDP) und Citidín-5'-Triffosphat (CTP) (CTP) (CTP) (CTP).
Desoxygenierte Äquivalente werden als Desoxicitidin-5'-monophosphat (DCMP), Desoxicitidin-5'-Diphosphat (DCDP) und Desoxicitidin-5'-Triffosphat (DCTP) bezeichnet.
Cytosin beteiligt sich in seinen verschiedenen Formen an verschiedenen Funktionen wie der Biosynthese von DNA und RNA, der Biosynthese von Glykoproteinen und der Regulation der Expression des Gens.
[TOC]
Struktur und Eigenschaften
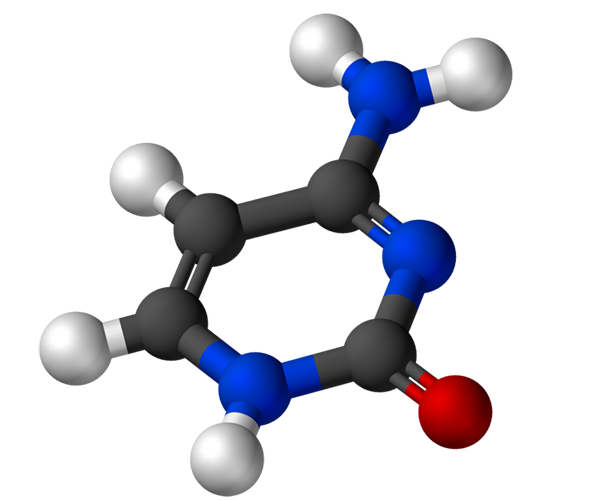
Das Cytosin, 4-Amino-2-Hydroxipirimidin, hat die empirische Formel C4H5N3Oder, dessen Molekulargewicht 111,10 g/mol beträgt und als weißes Pulver gereinigt wird.
Die Struktur des Zytosins ist ein heterocyclischer, aromatischer und planarer Ring. Die maximale Absorptionswellenlänge (ʎMax) Es ist 260 nm. Die Fusionstemperatur des Zytosins übersteigt 300 ºC.
Um ein Nukleotid zu bilden, bindet Cytosin kovalent durch Stickstoff 1 mit Mitteln. Carbon 5 'wird mit einer Phosphatgruppe verärgert.
Biosynthese
Die Pyrimidin -Nucleotid -Biosynthese hat einen gemeinsamen Weg, der aus sechs Schritten besteht, die durch Enzyme katalysiert werden. Die Straße beginnt mit Carbamoil-Phosphat-Biosynthese. In Prokaryoten gibt es nur ein Enzym: Carbamoil -Phosphat -Synthase. Dies ist verantwortlich für die Synthese von Pyrimidinen und Glutamin. In Eukaryoten gibt es Carbamoil -Phosphat -Synthase I und II, die für die Glutamin- und Pyrimidin -Biosynthese verantwortlich sind.
Der zweite Schritt besteht aus der Bildung von N-Carbamoilaspartato, aus Carboil-Phosphat und Aspartat, die durch das transpartat transcabamoilasa (ATCASA) reaktionsbedingt katalysiert werden.
Der dritte Schritt ist die Synthese von L-Dihydrotoat, das den Verschluss des Pyrimidinrings erzeugt. Dieser Schritt wird durch Dihydrootase katalysiert.
Der vierte Schritt ist die Bildung von Orotato, eine Redoxreaktion, die durch Dihydroorotatdehydrogenase katalysiert wird.
Der fünfte Schritt besteht aus der Bildung von Goldylat (OMP) unter Verwendung von Phosphoribosylpyrophosphat (PRPP) als Substrat und der Phosphoribosylorrotattransferase als Katalysator als Katalysator.
Der sechste Schritt ist die Bildung von Uridilaato (Uridin-5'-Monophosphat, Ump), die durch eine OMP-Decarboxylase katalysierte Reaktion.
Es kann Ihnen dienen: Flora und Fauna aus HidalgoDie folgenden Schritte bestehen aus der Phosphorylierung des UMP, die durch Kinasen katalysiert werden, um UTP zu bilden, und der Übertragung einer Aminogruppe von Glutamin zum UTP zur Bildung von CTP, die durch CTP -Synthetase katalysierte Reaktion.
Biosyntheseregulation
Bei Säugetieren erfolgt die Regulierung am Carbamoil -Phosphat -Synthase.
Carbamoil Syntasa II wird durch negatives Feedback reguliert. Seine Regulierungsbehörden, die UTP und der PRPP, sind Inhibitor und Aktivator dieses Enzyms.
In Geweben, die nicht hepatisch sind, ist Carbamoil -Phosphat -Synthase II die einzige Quelle für Carbamoil -Phosphat. Während in der Leber unter Bedingungen von überschüssigem Ammoniak produziert Carbamoil -Phosphat -Synthase I in den Mitochondrien Carbamoil -Phosphat, das zum Cytosol transportiert wird, wo es in den Weg der Biosynthese von Pyrimidinen eintritt.
Ein weiterer Regulationspunkt ist die OMP-Discarboxylase, die durch Wettbewerbshemmung reguliert wird. Das Produkt seiner Reaktion, UMP, konkurriert mit dem OMP für die Bindungsstelle in der OMP-Descarboxilasa.
Pyrimidine wie Cytosin werden recycelt
Das Recycling von Pyrimidinen hat als Funktion die Wiederverwendung von Pyrimidinen ohne die Notwendigkeit einer Novo -Biosynthese und vermeiden Sie den Abbauweg. Die Recyclingreaktion wird durch Phosphoribosyltransferase -Pyrimimidin katalysiert. Die allgemeine Reaktion ist wie folgt:
Pyrimidin + PRPP -> Nukleosidpyrimidin 5 '-Monophosphat + pPI
Bei Wirbeltieren findet sich Pysphoribosyltransferase -Pyrimimidin in Erythrozyten. Das Pyrimidine -Substrat dieses Enzyms sind Uracil, Timina und Orotato. Cytosin wird indirekt aus Uridin-5'-monophosphat recycelt.
Funktion in der DNA -Biosynthese
Während der DNA -Replikation werden die in der DNA enthaltenen Informationen in der DNA durch eine DNA -Polymerase kopiert.
Die RNA -Biosynthese benötigt Deoxynukleotide Tryphosphat (DNTP), nämlich: Dexitimid Trypphosphat (DTTP), Dexicitidie -Triffosphat (DCTP), DE -Alxyxyadenin Tryphyse (DATP) und Dexiguanin Tryphosphat (DTTP). Die Reaktion ist:
(DNA)n Abfall + DNTP -> (DNA)N+1 Abfall + PPI
Die Hydrolyse der anorganischen Pyrophosphat (PPI) liefert Energie für die Biosynthese der RNA.
Funktion bei der Stabilisierung der Struktur von DNA
In der doppelten DNUS -Helix ist ein Purin einer Kette durch Wasserstoffbrückenbindungen mit Pyrimidin der entgegengesetzten Kette verbunden. Somit ist Cytosin immer durch drei Wasserstoffbrückenbindungen mit Guanin verbunden: Adenin ist durch zwei Wasserstoffbrückenbindungen mit Timin verbunden.
Es kann Ihnen dienen: Bedeutung der BiologieWasserstoffbrückenbindungen werden gebrochen, wenn eine gereinigte native DNA -Lösung bei pH 7 Temperaturen von mehr als 80 ° C ausgesetzt ist. Dies führt dazu, dass der Doppel -DNA -Propeller zwei getrennte Ketten bildet. Dieser Prozess wird als Denaturierung bezeichnet.
Die Temperatur, bei der 50% der DNA denaturiert sind, ist als Schmelztemperatur (TM) bekannt. DNA -Moleküle, deren Guanin und Cytosin.
Das obige ist der experimentelle Test, dass eine größere Anzahl von Wasserstoffbrücken die nativen DNA -Moleküle besser stabilisiert.
Funktion von reichen Regionen in Cytosin in DNA
Kürzlich wurde festgestellt, dass die DNA des menschlichen Zellkerns interkalierte Motivstrukturen (IM) übernehmen kann. Diese Strukturen werden in Zytosinregionen produziert.
Die IM -Struktur besteht aus vier DNA -Ketten, im Gegensatz zur klassischen Doppelpropeller -DNA mit zwei Ketten. Insbesondere werden zwei parallele Duplexketten in eine antiparallelische Orientierung durchsetzt und durch ein Paar hemiprotoniertes Zytosinen zusammengehalten (C: C: C+).
Im menschlichen Genom finden sich IM -Strukturen in Regionen wie Promotoren und Telomeren. Die Anzahl der IM -Strukturen ist während der G1/S -Phase des Zellzyklus höher, in der die Transkription hoch ist. Diese Regionen sind Proteinerkennungsstellen, die an der Aktivierung von Transkriptionsmaschinen beteiligt sind.
Andererseits neigt die DNA in den Regionen, die reich an Guaninbasen (c) aufeinander folgen sind. Diese Form ist typisch für RNA- und doppelte DNA-Arn-Mischbänder während der Transkription und Replikation und zu bestimmten Zeiten, in denen DNA mit Protein verbunden ist.
Es wurde gezeigt, dass die Regionen mit aufeinanderfolgenden Zytosinbasen ein elektropositives Pflaster in der Hauptspalte der DNA erzeugen. Daher wird angenommen, dass diese Regionen an Proteine binden, die bestimmte genomische Regionen der genetischen Fragilität prädisponieren.
Funktion in der RNA -Biosynthese
Während des Transkripts werden die in der DNA enthaltenen Informationen in der RNA über eine PON -Polymerase -RNA kopiert. Die RNA -Biosynthese benötigt Tryposphat -Nukleoside (NTP), nämlich: Tryphyse Citidin (CTP), Tryposphat -Uridin (UTP), Adenin -Tryposphat (ATP) und Trypphosphat -Guanin (GTP). Die Reaktion ist:
Kann Ihnen dienen: Flora und Fauna von Campeche: Repräsentative Arten(RNA)n Abfall + NTP -> (RNA)N+1 Abfall + PPI
Die Hydrolyse der anorganischen Pyrophosphat (PPI) liefert Energie für die Biosynthese der RNA.
Funktion bei der Biosynthese von Glykoproteinen
Die sequentielle Übertragung von Hexosen zur Bildung der Oligosaccharide, O-Ligate zu Proteinen, erfolgt aus Nucleotid-Vorläufern.
In Wirbeltieren besteht der letzte Schritt der Biosynthese von Oligosacchariden von Oigosacchariden in der Zugabe von zwei Sinal-Säureabfällen (N-Acetylneuramin) aus einem Vorläufer von Citidin-5'-Monophosphat (CMP). Diese Reaktion wird im Transsack von Golgi erzeugt.
Cytosin- und Chemotherapie -Behandlungen gegen Krebs
Tetrahydropholatsäure (FH4) ist eine Gruppenquelle3, Und es ist für die DTMP -Biosynthese von Dump erforderlich. FH2 wird ebenfalls gebildet. Die FH2 -Reduktion auf FH4 erfordert eine Folsäure- und NADPH -Reduktase. Einige Folat -Reduktase -Inhibitoren wie Aminopterin und Metretothrexat werden bei Krebsbehandlungen verwendet.
Metotrexan ist ein wettbewerbsfähiger Inhibitor. Der Folatreduktase verbindet sich mit einer 100 -mal mehr Affinität zu diesem Inhibitor als zu seinem Substrat. Aminapterin wirkt ähnlich.
Die Hemmung von Folatreduktase behindert indirekt die DTMP -Biosynthese und damit die von DCTP. Direkte Hemmung erfolgt durch die Inhibitoren der Timidilat -Synthetase, die DTMP aus Dump katalysiert. Diese Inhibitoren sind 5-Fluorouracil und das 5-Fluor-2-Dioxiuridin.
Zum Beispiel ist 5-Fluoroacil kein Inhibitor, sondern zuerst zum Recyclingrouten im Desoxyurid-Mofosphat D (FDUMP), der an die Timidilat-Synthesase bindet und sie hemmt.
Substanzen analog zu Glutamin, Azaserin und Acivicin, hemmen Amidotransferase -Glutamin. Azarin war eine der ersten entdeckten Substanzen, die als selbstmörderisch inaktivend wirken.
Verweise
- Assi, h.ZU., Garavís, m., González, c., Und Damha, m.J. 2018. I-Motif-DNA: Strukturelle Merkmale und Bedeutung für die Zellbiologie. Kernsäurenforschung, 46: 8038-8056.
- Bohinski, r. 1991. Biochemie. Addison-Wesley Iberoamericana, Wilmington, Delaware.
- Devlin, t.M. 2000. Biochemie. Redaktionsreverté, Barcelona.
- Lodisch, h., Berk, a., Zipurski, s.L., Matsudaria, p., Baltimore, d., Darnell, J. 2003. Zell- und Molekularbiologie. Editorial Medica Panamericana, Buenos Aires, Bogotá, Caracas, Madrid, Mexiko, Sāo Paulo.
- Nelson, d. L., Cox, m. M. 2008. Lehninger-Prinzipien der Biochemie. W.H. Freeman, New York.
- Voet, d. und voet, j. 2004. Biochemie. John Wiley und Söhne, USA.

