Restriktionsenzyme Funktionen, Typen und Beispiele
- 4154
- 1317
- Frederike Birkemeyer
Der Restriktionsenzyme Sie sind Endonukleasen, die von bestimmten Bögen und Bakterien verwendet werden, um die Ausbreitung von Viren im Inneren zu hemmen oder zu "einschränken". Sie sind besonders häufig in Bakterien und Teil ihres fremden DNA -Verteidigungssystems, das als Einschränkung/Modifikationssystem bekannt ist.
Diese Enzyme katalysieren den doppelbandigen DNA -Schnitt an bestimmten Stellen, reproduzierbar und ohne zusätzliche Energie. Die meisten benötigen das Vorhandensein von Cofaktoren wie Magnesium oder anderen zweiwertigen Kationen, obwohl einige auch ATP- oder S-Adenosylmetionin benötigen.
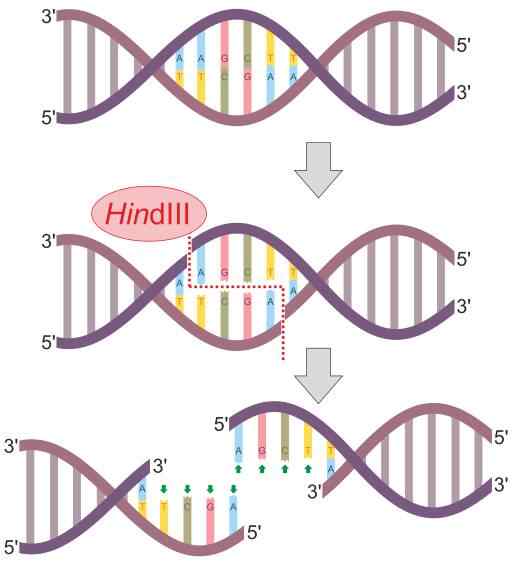
 Hindii-Restriktionsenzymreaktionsschema (Quelle: Helixitta [CC BY-SA 4.0 (https: // creativecommons.Org/lizenzen/by-sa/4.0)] über Wikimedia Commons)
Hindii-Restriktionsenzymreaktionsschema (Quelle: Helixitta [CC BY-SA 4.0 (https: // creativecommons.Org/lizenzen/by-sa/4.0)] über Wikimedia Commons) Die Beschränkungsendonukleasen wurden 1978 von Daniel Nathans, Arber Werner und Hamilton Smith entdeckt, der den Nobelpreis für Medizin für ihre Entdeckung erhielt. Sein Name leitet sich im Allgemeinen aus dem Organismus ab, wo sie zuerst beobachtet werden.
Solche Enzyme werden häufig bei der Entwicklung von DNA -Klonierungsmethoden und anderen Strategien für molekulare Biologie und Gentechnik eingesetzt. Seine Eigenschaften der Erkennung spezifischer Sequenzen und die Schnittkapazität der Sequenzen in der Nähe der Erkennungsstellen machen sie leistungsstarke Werkzeuge in genetischen Experimenten.
Die von den in einem bestimmten DNA -Molekül wirksamen Restriktionsenzymen erzeugten Fragmente können verwendet werden.
Einige Restriktionsenzyme können die gleiche Erkennungsstelle in der DNA haben, aber sie schneiden sie nicht unbedingt auf die gleiche Weise ab. Daher gibt es Enzyme, die Schnitte machen, die Romos und Enzyme hinterlassen, die die Verlassen der zusammenhängenden Extreme schneiden, die unterschiedliche Anwendungen in der Molekularbiologie haben.
Derzeit gibt es Hunderte verschiedener Einschränkungsenzyme im Handel, die von verschiedenen Handelshäusern angeboten werden. Diese Enzyme fungieren als "maßgeschneiderte" molekulare Schere für verschiedene Zwecke.
[TOC]
Funktionen
Restriktionsenzyme erfüllen die entgegengesetzte Funktion von Polymerasen, da sie hydrolysiert oder die Esterbindung innerhalb der Phosphodiéster -Bindung zwischen benachbarten Nukleotiden in einer Nukleotidkette durchbricht.
In der molekularen Biologie und Gentechnik sind sie weit verbreitete Werkzeuge zur Konstruktion von Expressions- und Klonierungsvektoren sowie zur Identifizierung spezifischer Sequenzen. Sie sind auch nützlich für die Konstruktion rekombinanter Genome und haben ein großes biotechnologisches Potenzial.
Jüngste Fortschritte in der Gentherapie verwenden den aktuellen Einsatz von Restriktionsenzymen zur Einführung von Genen, die in Vektoren ermittelt werden, die Fahrzeuge für den Transport solcher Gene in lebende Zellen sind, und dass sie wahrscheinlich die Fähigkeit haben, sich in das Zellgenom einzusetzen, um dauerhafte Veränderungen durchzuführen, um dauerhafte Veränderungen durchzuführen.
Wirkmechanismus
Restriktionsenzyme können den Doppelband -DNA -Schnitt katalysieren, obwohl einige in der Lage sind, einfache Band -DNA -Sequenzen und sogar RNA zu erkennen. Der Schnitt tritt nach der Erkennung der Sequenzen auf.
Kann Ihnen dienen: Glioxylatzyklus: Eigenschaften, Reaktionen, Regulierung, FunktionenDer Wirkmechanismus besteht aus der Hydrolyse des Phosphodiéster -Zusammenhangs zwischen einer Phosphatgruppe und einer Desoxyribose im Skelett jeder DNA von DNA. Viele der Enzyme können an den gleichen Ort schneiden, den sie erkennen, während andere vor oder nach demselben zwischen 5 und 9 Basispaaren schneiden.
Normalerweise schneiden diese Enzyme am 5' -Ende der Phosphatgruppe, wodurch DNA -Fragmente mit einem 5'.
Da Proteine nicht direkt in den direkten Kontakt mit der DNA -Erkennungsstelle kommen, müssen diese aufeinanderfolgenden Zeiten übersetzt werden.
Während des enzymatischen Schnitts befindet sich die Phosphodiéster -Verbindung der einzelnen DNA -Stränge in einem der aktiven Stellen von Restriktionsenzymen. Wenn das Enzym die Erkennungs- und Schnittstelle verlässt.
Leute
Derzeit sind fünf Arten von Einschränkungssenzymen bekannt. Als nächstes eine kurze Beschreibung jedes einzelnen:
Typ -I -Beschränkungsenzyme
Diese Enzyme sind große Pentamerproteine mit drei Untereinheiten, einer Einschränkung, einer Methylierung und einer anderen für die Erkennung von DNA -Sequenzen. Diese Endonukleasen sind multifunktionale Proteine, die in der Lage sind, Einschränkungen und Modifikationsreaktionen zu katalysieren, ATPASA- und auch DNA -Topoisomera -Aktivitäten aufweisen.
Die Enzyme dieser Art waren die ersten Endonukleasen, die entdeckt wurden, sie wurden zum ersten Mal in den 1960er Jahren gereinigt und seitdem wurden sie mit großer Tiefe untersucht.
Typ -I -Enzyme werden nicht weit verbreitet als biotechnologisches Werkzeug, da sich die Schneidstelle in einem variablen Abstand von bis zu 1 befinden kann.000 Basispaare bezüglich der Erkennungsstelle, was sie in Bezug auf die experimentelle Reproduzierbarkeit unzuverlässig macht.
Typ -II -Restriktionsenzyme
Sie sind Enzyme, die aus Homodimeren oder Tetrameren bestehen, die die DNA in Orte schneiden, die zwischen 4 und 8 bp Länge definiert sind. Diese Schneidstellen sind typischerweise palindrom, dh sie erkennen Sequenzen, die in beide Richtungen auf die gleiche Weise gelesen werden.
Viele von Typ -II -Restriktionsenzymen in Bakterien schneiden die DNA, wenn sie ihren Fremdcharakter erkennen, da sie nicht die typischen Modifikationen haben, die die eigene DNA haben sollte.
Dies sind einfachere Einschränkungsenzyme, da sie keinen anderen Cofaktor als Magnesium (Mg+) benötigen, um die DNA -Sequenzen zu erkennen und zu schneiden.
Die Präzision der Typ -II -Restriktionsenzyme beim Erkennen und Schneiden einfacher Sequenzen in DNA in genauen Positionen macht sie zu einem der am häufigsten verwendeten und unverzichtbaren in den meisten Zweigen der Molekülbiologie.
Kann Ihnen dienen: Mutualismus: Eigenschaften, Typen, BeispieleInnerhalb der Gruppe von Typ -II -Restriktionsenzymen sind mehrere Unterklassen, die nach bestimmten Eigenschaften klassifiziert sind, die für jeden einzigartig sind. Die Klassifizierung dieser Enzyme wird durch Hinzufügen von Buchstaben des Alphabets von A bis Z durchgeführt, die dem Namen des Enzyms folgen,.
Einige der bekanntesten Unterklassen für ihre Nützlichkeit sind:
Unterklasse IIA
Sie sind unterschiedliche Untereinheiten Dímeros. Sie erkennen asymmetrische Sequenzen und werden als ideale Vorläufer für die Erzeugung von Schneidenzymen verwendet.
Subklasse IIB
Sie bestehen aus einem weiteren Dimeren und schneiden die DNA auf beiden Seiten der Erkennungssequenz ab. Sie schneiden beide DNA -Stränge später in einem Basispaar -Intervall aus der Erkennungsstelle aus.
IIC -Unterklasse
Enzyme dieser Art sind Polypeptide mit Teilung und Modifikationsfunktionen von DNA -Strängen. Diese Enzyme schneiden beide Stränge asymmetrisch ab.
Unterklasse Iie
Die Enzyme dieser Unterklasse sind am häufigsten in der Gentechnik verwendet. Sie haben eine katalytische Stelle und erfordern im Allgemeinen einen Alestro -Effektor. Diese Enzyme müssen mit zwei Kopien ihrer Erkennungssequenz interagieren, um ein effizientes Schneiden zu machen. Innerhalb dieser Unterklasse befinden sich Ecorii- und Ecori -Enzyme.
Typ -III -Restriktionsenzyme
Die Endonukleasen vom Typ III III besteht nur aus zwei Untereinheiten, einer ist für die Erkennung und Modifikation von DNA verantwortlich, während der andere für den Schnitt der Sequenz verantwortlich ist.
Diese Enzyme benötigen zwei Cofaktoren für ihren Betrieb: ATP und Magnesium. Die Restriktionsenzyme dieser Art haben zwei asymmetrische Erkennungsstellen, translozieren Sie die DNA in ATP -abhängiger Weise und schneiden Sie sie zwischen 20 und 30 bp neben der Erkennungsstelle ab.
Typ -IV -Restriktionsenzyme
Typ -IV -Enzyme sind leicht zu identifizieren, da sie DNA mit Methylierungsspuren schneiden und aus verschiedenen Untereinheiten bestehen, die für das Erkennen und Schneiden der DNA -Sequenz verantwortlich sind. Diese Enzyme verwenden GTP -Cofaktoren und zweiwertige Magnesiumium.
Spezifische Schneidstellen umfassen Nukleotidketten mit methylierten oder hydroxymetyblydulierten Cytosinresten in einem oder beiden Strängen von Nukleinsäuren.
Typ -V -Restriktionsenzyme
Diese Klassifizierungsgruppe gruppiert Crisper-Cas-Typ-Enzyme, die spezifische DNA-Sequenzen von Eindringorganismen identifizieren und schneiden. CAS -Enzyme verwenden einen RNA -Strang, der synthetisierte Leitfaden für knirschigere, eindringende Organismen erkennen und angreift.
Enzyme, die als Typ V klassifiziert sind. Sie können DNA. Seine Flexibilität und einfache Beschäftigung machen diese Enzyme zu einem der am häufigsten verwendeten Werkzeuge in der Gentechnik zusammen mit Typ -II -Enzymen.
Kann Ihnen dienen: PropionibacteriumBeispiele
Restriktionsenzyme wurden zum Nachweis von DNA.
Gegenwärtig haben die zur Transformation von Bakterien mit verschiedenen Zwecken verwendeten Vektoren Multiclonage -Stellen, an denen Erkennungsstellen für mehrere Restriktionsenzyme gefunden werden.
Unter diesen Enzymen sind die beliebtesten Ecori, II, III, IV und V, die erstmals erhalten und beschrieben wurden UND. coli; Hindiii, von H. Influenzae und Bamhi von B. Amyloliquefaciens.
Verweise
- Bikle, t. ZU., & Kruger, D. H. (1993). Biologie der DNA -Einschränkung. Mikrobiologische Bewertungen, 57(2), 434-450.
- Boyaval, p., Moineau, s., Romero, d. ZU., & Horvath, p. (2007). CRISPR liefert eine Bekanntschaft mit Viren in Prokaryoten. Wissenschaft, 315(März), 1709-1713.
- Goodsell, d. (2002). Die molekulare Perspektive: Restriktionsendonukleasen. Stammzellen Grundlagen der Krebsmedizin, zwanzig, 190-191.
- Halford, s. UND. (2001). Hüpfen, Springen und Schleifen durch Restriktionsenzyme. Transaktionen der biochemischen Gesellschaft, 29, 363-373.
- Jeltsch, a. (2003). Aufrechterhaltung der Speziesidentität und Kontrolle der Speziation von Bakterien: eine neue Funktion für Einschränkungen/Modifikationssysteme? Gen, 317, 13-16.
- Krebs, j., Goldstein, e., & Kilpatrick, s. (2018). Lewins Gene XII (12 ed.). Burlington, Massachusetts: Jones & Bartlett Learning.
- Li, und., Brot, s., Zhang und., Ren, m., Feng, m., Peng, n.,... sie, q. (2015). CRISPR-CAS-Systeme vom Typ I und Typ III zur Genombearbeitung nutzen. Nukleinsäurenforschung, 1-12.
- Loenen, w. ZU. M., Dryden, d. T. F., Raleight, e. ZU., & Wilson, G. G. (2013). Einschränkungsenzyme von Typ I und ihre Verwandten. Nukleinsäurenforschung, 1-25.
- Nathans, d., & Smith, h. ENTWEDER. (1975). Restriktionsendonukleasen in der Analyse und Umstrukturierung von DNA -Molekülen. Annu. Rev. Biochem., 273-293.
- Nei, m., & Tajima, f. (1981). DNA -Polymorphismus, die durch Restriktionsendonukleasen nachweisbar sind. Genetik, 145-163.
- Pingoud, a., Fuxreiter, m., Pingoud, v., & Wende, w. (2005). Zell- und molekulare Biowissenschaften Typ -II -Restriktionendonukleasen: Struktur und Mechanismus. CMLS -Zell- und molekulare Biowissenschaften, 62, 685-707.
- Roberts, r. (2005). Wie Einschränkungenenzyme zu Arbeitspuren der molekularen Biologie wurden. PNAs, 102(17), 5905-5908.
- Roberts, r. J., & Murray, k. (1976). Restriktionsendonukleasen. Kritische Bewertungen in der Biochemie, (November), 123-164.
- Stoddard, geb. L. (2005). Struktur und Funktion endonuklease nach Hause. Vierteljährliche Bewertungen der Biophysik, 1-47.
- Teck, m. R., & Dryden, D. T. F. (2005). Die Biologie der Einschränkung und der Anti-Restriktion. Aktuelle Meinung in der Mikrobiologie, 8, 466-472. https: // doi.org/10.1016/j.Mein b.2005.06.003
- Wilson, g. G., & Murray, n. UND. (1991). Einschränkungen und Modifikationssysteme. Annu. Rev. Genet., 25, 585-627.
- Wu, z., & Mou, k. (2016). Genomische Einblicke in die Campylobacter Jejuni -Virulenz und die Populationsgenetik. INFEC. Dis. Übersetzt. Med., 2(3), 109-19.
- Yuan, r. (1981). Struktur und Mechanismus multifunktionaler Restriktionsendonukleasen. Annu. Rev. Biochem., fünfzig, 285-315.
- « Generation von Merkmalen, Autoren und Arbeiten von 1914
- Gegenseitig ausschließende Ereignisse Eigenschaften und Beispiele »

