Haplootyp -Studienmethoden, Diagnose, Krankheiten
- 4657
- 1031
- Lewis Holzner
A Haploootype Es ist eine Region des Genoms, die dazu neigt, über mehrere Generationen zusammen zu erben. Normalerweise befindet sich alles im selben Chromosom. Haplootypen sind das Produkt der genetischen Bindung und bleiben während der genetischen Rekombination intakt.
Das Wort "Haplotipo" stammt aus einer Kombination aus dem Wort "haploid" und dem Wort "Genotyp". "Haploid" bezieht sich auf Zellen mit einer einzigen Reihe von Chromosomen und "Genotyp" spricht über die genetische Zusammensetzung eines Organismus.
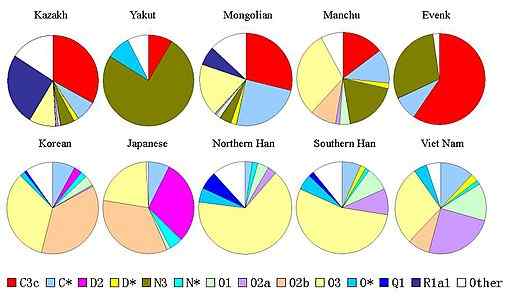
 Schema der Verteilung von Chromosomen -Haplotypen und in asiatischen Populationen (Quelle: Moogalord [Public Domain] über Wikimedia Commons nach der Definition kann ein Haplotyp ein paar oder mehr Gene beschreiben, die zusammen auf einem Chromosom von einem elterlichen oder elterlich geerbt werden, oder mehr Kann ein Chromosom beschreiben, das vollständig von einem Eltern geerbt wird, wie es bei Chromosom und Männern der Fall ist.
Schema der Verteilung von Chromosomen -Haplotypen und in asiatischen Populationen (Quelle: Moogalord [Public Domain] über Wikimedia Commons nach der Definition kann ein Haplotyp ein paar oder mehr Gene beschreiben, die zusammen auf einem Chromosom von einem elterlichen oder elterlich geerbt werden, oder mehr Kann ein Chromosom beschreiben, das vollständig von einem Eltern geerbt wird, wie es bei Chromosom und Männern der Fall ist.
Wenn Haplotypen beispielsweise Gene für zwei verschiedene phänotypische Merkmale wie Haarfarbe und Augenfarbe teilen, werden Individuen mit Haarfarben auch das andere Gen für die Farbe der Augen besitzen.
Haploootypen sind heute eines der am häufigsten verwendeten Werkzeuge für die Untersuchung der Genealogie, um den Ursprung von Krankheiten zu verfolgen und die genetische Variabilität und Phylogeographie von Populationen verschiedener Arten von Lebewesen zu charakterisieren.
Es gibt mehrere Werkzeuge für die Untersuchung von Halootypen, eines der heutigen am häufigsten verwendeten. "Haplotyp -Karte„(HAPMAP), eine Webseite, auf der die Genomsegmente sind, die Haplotypen sind.
[TOC]
Studienmethoden
Haploootypen stellen eine Gelegenheit dar, die Erbe von Genen und ihren Polymorphismus zu verstehen. Mit der Entdeckung der Technik "Polymerase -Kettenreaktion" (PCR, English "Polymerase Kettenreaktion”) Er hat sich in der Studie von Haplotypen weit verbreitet.
Derzeit gibt es zahlreiche Methoden für die Untersuchung von Haplotypen, einige der meisten Highlights sind:
DNA -Sequenzierung und Einzelnukleotidpolymorphismus -Nachweis (SNPs)
Die Entwicklung von Sequenzierungstechnologien für die neue Generation war ein großer Sprung für die Untersuchung von Haplotypen. Neue Technologien ermöglichen Variationen von bis zu einer einzelnen Nukleotidbase in bestimmten Regionen eines Haplotyps.
Kann Ihnen dienen: rekombinante DNA: Technik, Anwendungen und FundamenteIn Bioinformatik wird der Begriff Haplotyp auch verwendet, um auf die Vererbung eines einzelnen Nukleotidpolymorphismen (SNP) in DNA -Sequenzen zu beziehen.
Die Kombination von Bioinformatikprogrammen mit der Erkennung von Haplotypen unter Verwendung der Sequenzierung neuer Generation kann genau identifiziert werden, die Position, den Austausch und die Wirkung der Veränderung jeder Basis auf dem Genom einer Population.
Mikrosatelliten (SSRs)
Die Mikrosatelliten oder SSRs leiten ihren Namen aus dem Englischen ab “SWiederholung implementieren Und Kurze Tandem -Wiederholung". Dies sind kurze Nukleotidsequenzen, die nacheinander in einer Genomregion wiederholt werden.
Es ist üblich, Mikrosatelliten innerhalb der nicht kodierenden Haplotypen zu finden, daher können durch den Nachweis von Variationen der Anzahl der Wiederholungen von Mikrosatelliten unterschiedliche Allele in den Haplotypen von Individuen beobachtet werden.
Mikrosatellitenmolekulare Marker wurden zum Nachweis von endlosen Haplotypen aus dem Sexat von Pflanzen wie Papaya (Papaya () entwickeltCarica Papaya) bis zur Erkennung menschlicher Krankheiten wie Falciform Anämie.
Polymorphismen der amplifizierten Fragmente (AFLP)
Diese Technik kombiniert Amplifikation mit PCR -Reaktionen mit DNA -Verdau mit zwei verschiedenen Restriktionsenzymen. Die Technik erkennt polymorphe Loci in Haplootypen gemäß den verschiedenen Schneidstellen in der DNA -Sequenz.
Um die Technik besser zu veranschaulichen, stellen wir uns drei Stofffragmente derselben Länge vor, aber an verschiedene Orte geschnitten (diese Fragmente repräsentieren drei Fragmente amplifizierter Haplotypen unter Verwendung der PCR -Technik).
Zum Zeitpunkt des Schnitts des Stoffes werden viele Teile unterschiedlicher Größe erhalten, da jedes Tuch an verschiedenen Stellen geschnitten wird. Bei der Bestellung der Fragmente nach der Art von Stoff, aus der sie kommen, können wir beobachten, wo die Unterschiede zwischen Stoffen oder Haplotypen gefunden werden.
Diagnostik und Krankheiten
Ein wichtiger Vorteil der genetischen Untersuchung von Haplotypen besteht darin, dass diese für Tausende von Generationen fast intakt oder unverändert bleiben, und dies ermöglicht die Identifizierung von entfernten Vorfahren und jede der Mutationen, mit denen Individuen zur Entwicklung von Krankheiten beitragen.
Kann Ihnen dienen: phänotypische VariationenHapoPootypen in der Menschheit variieren je nach Rassen, und aus dieser Kugel wurden Gene in Haplotypen nachgewiesen, die in jedem der menschlichen Rassen schwere Krankheiten verursachen.
Im Projekt Hapmap Es sind vier Rassengruppen enthalten: Europäischer, nigerianischer Yoruba, chinesisches Han und Japanisch.
Auf diese Weise das Projekt Hapmap Es kann die verschiedenen Gruppen von Populationen abdecken und den Ursprung und die Entwicklung vieler erblicher Krankheiten verfolgen, die jedes der vier Rennen betreffen.
Eine der Krankheiten, die am häufigsten unter Verwendung einer Haplotypanalyse diagnostiziert wird, ist die Falciform -Anämie beim Menschen. Diese Krankheit wird diagnostiziert, indem die Häufigkeit afrikanischer Haplotypen zu einer Population verfolgt wird.
Wenn Sie eine Krankheit aus Afrika sind, können Sie Menschen, die die Mutation in der genetischen Sequenz für die Beta -Globinas von Erythrozyten in Form eines HOZ (charakteristisch für die Pathologie) besitzen (charakteristisch für die Pathologie), leicht verfolgen, wenn Sie eine Krankheit aus Afrika stammen, die sie ursprünglich aus Afrika identifizieren, und die Identifizierung von afrikanischen Haplotypen (charakteristisch für die Pathologie) problemlos verfolgt.
Beispiele
Mit Haplotypen phylogenetische Bäume, die evolutionäre Beziehungen zwischen jedem der Haplotypen darstellen.
Einer der am meisten untersuchten Zweige durch Haplotypen ist die Entwicklung des Menschen immunsystem. Haploootypen, die für den Rezeptor vom Typ des Tolls (eine Schlüsselkomponente des angeborenen Immunsystems) für das Genom Neandertaler und Denisovanos identifiziert wurden.
Dies ermöglicht es ihnen zu verfolgen, wie sich genetische Sequenzen in den Populationen von "modernen" Menschen in Bezug auf die Haplotyp -Sequenzen verändert haben.
Aufbau eines Netzwerks genetischer Beziehungen aus dem mitochondrialen Haplotyp.
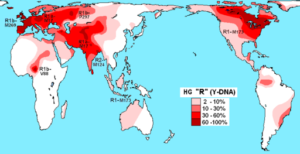 Haplogruppenverteilung R (DNA-y) in nativen Populationen (Quelle: Maucioni [CC von 3.0 (https: // creativecommons.Org/lizenzen/bis/3.0)] über Wikimedia Commons) Die Vielfalt der Haplotypen wird verwendet, um die genetische Vielfalt der in Gefangenschaft erhobenen Tiere zu verfolgen und zu untersuchen. Diese Techniken werden speziell für schwierige Arten verwendet, um Wildtiere zu überwachen.
Haplogruppenverteilung R (DNA-y) in nativen Populationen (Quelle: Maucioni [CC von 3.0 (https: // creativecommons.Org/lizenzen/bis/3.0)] über Wikimedia Commons) Die Vielfalt der Haplotypen wird verwendet, um die genetische Vielfalt der in Gefangenschaft erhobenen Tiere zu verfolgen und zu untersuchen. Diese Techniken werden speziell für schwierige Arten verwendet, um Wildtiere zu überwachen.
Tierarten wie Haie, Vögel und große Säugetiere wie Jaguare, unter anderem Elefanten werden ständig durch mitochondriale Haplotypen genetisch bewertet, um den genetischen Zustand der Populationen in Gefangenschaft zu überwachen.
Verweise
- Bahlo, m., Stankovich, j., Geschwindigkeit, t. P., Rubio, j. P., Burfoot, r. K., & Foot, s. J. (2006). DEGTEINIEREN SIE GENOME WEITER HAPLOTYP TRIGUNG mithilfe von SNP- oder Mikrosatelliten -Haplotyp -Daten. Human Genetics, 119 (1-2), 38-50.
- Dannemann, m., Andrés, a. M., & Kelso, j. (2016). Introgress von Neandertaler-Ando-Denisovan-ähnlichen Haplotypen trägt zu einer adaptiven Variation bei menschlichen gebührenähnlichen Empfängern bei. Das American Journal of Human Genetics, 98 (1), 22-33.
- Von vries, h. G., Van der meulen, m. ZU., Rozen, r., Halley, d. J., Scheffer, h., Leo, p.,… & Du Meerman, G. J. (neunzehn sechsundneunzig). Haplotyp -Identität zwischen Personen, die ein CFTR -Mutations -Allel teilen. Human Genetics, 98 (3), 304-309
- Schlucken, m. ZU., Leaver, a. L., Christiansen, f. T., Witt, c. S., Abraham, l. J., & Dawkins, r. L. (1992). Haplotypen der Ahnen: Konservatische Population MHC -Haplotypen. Menschliche Immunologie, 34 (4), 242-252.
- Fellows, m. R., Hartman, t., Hermelin, d., Landau, g. M., Rosamond, f., & Rozenberg, L. (2009, Juni). Haplotyp -Inferenz durch plausible Haplotyp -Daten eingeschränkt. Im jährlichen Symposium zum kombinatorischen Muster -Matching (PP. 339-352). Springer, Berlin, Heidelberg.
- Gabriel, s. B., Schaffner, s. F., Nguyen, h., Moore, j. M., Roy, j., Blumenstiel, geb.,... & liu-cordero, s. N. (2002). Die Struktur von Haplotypblöcken im menschlichen Genom. Science, 296 (5576), 2225-2229.
- Internationales Hapmap -Konsortium. (2005). Eine Haplotypkarte des menschlichen Genoms. Nature, 437 (7063), 1299.
- Wynne, r., & Wilding, c. (2018). Mithochondriale DNA -Haplotyp -Vielfalt und Herkunft von Gefangenen Sand -Tigerhaie (Carcharias Taurus). Journal of Zoo and Aquarium Research, 6 (3), 74-78.
- Yoo, und. J., Tang, j., Kaslow, r. ZU., & Zhang, k. (2007). Haplotyp-Inferenz für vorliegende Genotypdaten unter Verwendung früherer identifizierter Haplotypen und Haplotyp-Muster. Bioinformatik, 23 (18), 2399-2406.
- Jung, n. S. (2018). Aplastische Anämie. The New England Journal of Medicine, 379 (17), 1643-1656.
- « Hollandische Vererbungseigenschaften, Funktionen von Genen, Degeneration
- Lentén größere Merkmale, Lebensraum, Eigenschaften, Pflege »

