Übersetzung des DNA -Prozesses in Eukaryotas und Prokaryoten
- 3399
- 77
- Said Ganzmann
Der DNA -Übersetzung Es ist der Prozess, durch den die Informationen, die in den während des Transkripts erstellten Boten enthalten sind.
Aus der zellulären Perspektive ist die Expression eines Gens eine relativ komplexe Angelegenheit, die in zwei Schritten auftritt: Transkription und Translation.
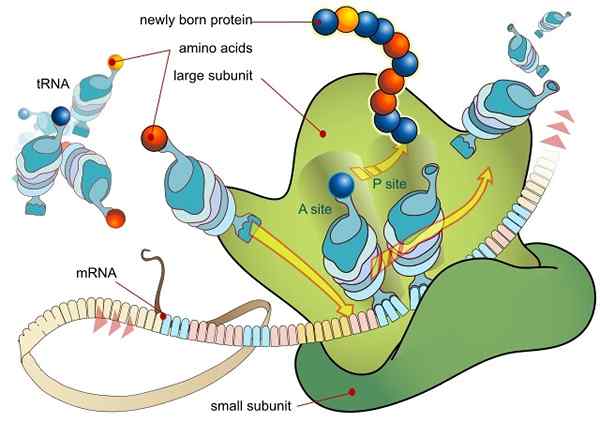
 RNA -Translation durch ein Ribosom (Quelle: Ladyofhats / Public Domain, über Wikimedia Commons)
RNA -Translation durch ein Ribosom (Quelle: Ladyofhats / Public Domain, über Wikimedia Commons) Alle Gene, die exprimiert werden (unabhängig davon.
Das Transkript wird durch spezielle Enzyme erreicht, die als RNA.
Für Gene, die Proteine codieren, werden die in reifen RNM enthaltenen Informationen „gelesen“ und in Aminosäuren gemäß dem genetischen Code übersetzt, was angibt, welches Codon- oder Nukleotid -Triplett jeder bestimmten Aminosäure entspricht.
Die Spezifikation der Aminosäuresequenz eines Proteins hängt daher von der anfänglichen Sequenz von Stickstoffbasen in der DNA ab, die dem Gen und dann in der RNA entspricht, die diese Informationen vom Kern zum Cytosol (in eukaryotischen Zellen) transportiert. Prozess, der auch als die Synthese von Protein, die von RNM geleitet wird, definiert ist.
Angesichts der Tatsache, dass es 64 mögliche Kombinationen der 4 Stickstoffbasen gibt, die DNA und RNA bilden, und nur 20 Aminosäuren, kann eine Aminosäure von verschiedenen Tripletts (Codons) codiert werden, so dass der genetische Code ist. " degeneriert "(mit Ausnahme des Aminosäuremetionins, das durch ein einzigartiges Aug -Codon codiert wird).
[TOC]
Eukaryota-Übersetzung (Stufenprozesse)
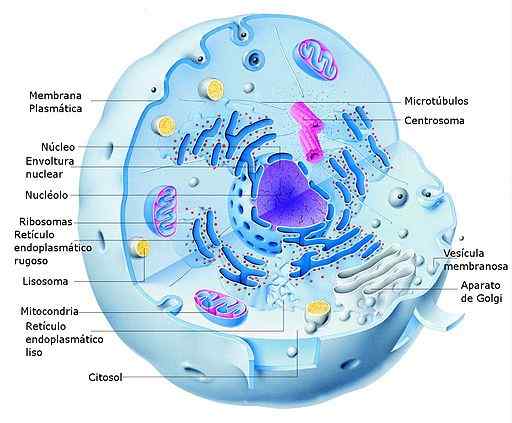
 Diagramm einer tierischen Eukaryot -Zelle und ihrer Teile (Quelle: Alejandro Porto [CC0] über Wikimedia Commons)
Diagramm einer tierischen Eukaryot -Zelle und ihrer Teile (Quelle: Alejandro Porto [CC0] über Wikimedia Commons) In eukaryotischen Zellen findet die Transkription im Kern und in der Translation in das Cytosol statt, so dass die RNMs, die während des ersten Prozesses gebildet werden ).
Es ist wichtig zu erwähnen, dass die Kompartimentierung von Transkription und Translation in Eukaryoten für den Kern gilt, aber für Organellen mit ihrem eigenen Genom wie Chloroplasten und Mitochondrien nicht dasselbe ist, die ähnliche Systeme wie prokaryotische Organismen haben.
Eukaryotische Zellen haben auch zytosolische Ribosomen, die an den Membranen des endoplasmatischen Retikulums (raues endoplasmatisches Retikulum) gebunden sind.
- RNM -Verarbeitung vor ihrer Übersetzung
RNMs werden an ihren Enden modifiziert, wenn sie transkribiert werden:
- Wenn das 5'-Ende des RNM während der Transkription aus der Oberfläche der RNA-Polymerase II entsteht, wird dies sofort von einer Gruppe von Enzymen "angegriffen" Nukleotid des RNM durch eine 5 'Triffosphatverbindung, 5'.
Kann Ihnen dienen: Codon- Das 3' -Ende des RNM erleidet eine "Clivaje" durch eine Endonuklease, die eine kostenlose Hydroxylgruppe 3 'erzeugt Zeit für ein Enzym Poly (A) Polymerase.
Die "Haube 5 '" und der "Schwanz Poly A ”Sie erfüllen Funktionen beim Schutz von RNM -Molekülen vor Abbau und außerdem arbeiten sie beim Transport von reifen Transkripten zum Cytosol bzw. in der Einleitung und Beendigung der Übersetzung.
COrte und Empalme
Nach der Transkription gehen die „primären“ RNMs mit ihren beiden modifizierten Extremen, die noch im Kern vorhanden sind, durch einen „Schnitt- und Spleißprozess“, durch den Introniksequenzen im Allgemeinen eliminiert werden und die resultierenden Exons verbunden sind (postregionale Verarbeitung), Mit den reifen Transkripten, die den Kern verlassen und das Cytosol erreichen.
Der Schnitt und das Spleißen werden von einem riboprotischen Komplex ausgeführt, der als der genannt wird Esplicleosoma (Anglizität von Spliceosom), gebildet durch fünf kleine Ribonukleoproteine und RNA -Moleküle, die die Regionen, die aus dem primären Transkript eliminiert werden müssen.
In vielen Eukaryoten gibt es ein Phänomen, das als „Schneiden und alternatives Gelenk“ bezeichnet wird, was bedeutet, dass verschiedene Arten von postregistrierenden Modifikationen unterschiedliche oder Isoenzymproteine verursachen können, die sich in einigen Aspekten ihrer Sequenzen voneinander unterscheiden.
- Die Ribosomen
Wenn reife Transkripte den Kern verlassen und zur Translation in Cytosol transportiert werden, werden diese durch den als Ribosom bekannten Translationskomplex verarbeitet, der aus einem Proteinkomplex besteht, der mit RNA -Molekülen assoziiert ist.

Die Ribosomen bestehen aus zwei Untereinheiten, einer "groß" und einem "kleinen", die im Cytosol frei dissoziiert sind und sich mit dem mRNA -Molekül, das übersetzt.
Die Vereinigung zwischen den Ribosomen und der mRNA hängt von spezialisierten RNA.
Die ARNT sind molekulare "Adapter", denn durch eines ihrer Enden können sie "jedes Codon oder Triplett in der reifen RNA (durch Komplementarität von Basen) lesen, und durch das andere können sie sich der Aminosäure anschließen, die durch das Codon" gelesen "codiert ist.
RNR -Moleküle hingegen sind für die Beschleunigung (Katalyse) des Bindungsprozesses jeder Aminosäure in der entstehenden Peptidkette verantwortlich.
Ein eukaryotischer reifen RNM kann von vielen Ribosomen "gelesen" werden, so oft wie die Zelle es anzeigt. Mit anderen Worten, das gleiche RNM kann zu vielen Kopien desselben Proteins führen.
Startcodon und Leserahmen starten
Wenn eine reife RNM von den ribosomalen Untereinheiten nähert, "scannen" die Sequenz des Molekül.
Es kann Ihnen dienen: Monoploidie: Wie es passiert, Organismen, Frequenz und NutzenDas AG -Codon definiert den Leserahmen für jedes Gen und außerdem die erste Aminosäure aller Proteine, die in die Natur übersetzt werden.
Kündigungscodons
Drei weitere Codons wurden als solche identifiziert, die Übersetzungsabschlüsse induzieren: UAA, UAG und UGA.
Diese Mutationen, die eine Änderung der Stickstoffbasen im Triplett implizieren, die für eine Aminosäure codiert und die zu Terminierungscodons führen.
Nicht übertriebene Regionen
Nahe dem 5 'Ende der reifen RNM -Moleküle gibt es Regionen, die nicht übersetzt werden (UTR, vom Englischen Untranslate Region), auch als „Leader“ -Sequenzen bezeichnet, die sich zwischen dem ersten Nukleotid und dem Beginn der Übersetzung befinden (August).
Diese UTR -Regionen, die nicht übersetzt werden, haben spezifische Stellen für die Vereinigung mit Ribosomen und Menschen. Sie haben beispielsweise eine ungefähre Länge von 170 Nukleotiden, unter denen es regulatorische Regionen gibt, Proteinbindungsstellen, die bei der Regulierung der Translation usw. funktionieren, usw.
- Übersetzungsbeginn
Die Übersetzung sowie die Transkription besteht aus 3 Phasen: einer der Initiation, einer anderen der Verlängerung und schließlich einer der Kündigungen.
Einleitung
Es besteht aus der Versammlung des Translationskomplexes auf dem RNM, der die Vereinigung von drei Proteinen verdient Initiationsfaktor) IF1, IF2 und IF3 zur kleinen Untereinheit des Ribosoms.
Der durch die Initiationsfaktoren und die kleine ribosomale Untereinheit gebildete "vor -initiation" -Komplex werden wiederum mit einem ARN.
Diese Ereignisse führen zur RNM -Vereinigung mit der großen ribosomalen Untereinheit, die zur Freisetzung von Initiationsfaktoren führt. Die große Untereinheit des Ribosoms verfügt über 3 Gewerkschaftsstellen für ARNT -Moleküle: Standort A (Aminosäure), Standort P (Polypeptid) und Standort E (Ausgang).
Site A schließt sich dem Aminoacil-Arnt-Anticod an, der zu dem der übersetztes mRNA ergänzt wird; In der P -Stelle wird die Aminosäure vom ARNT auf das entstehende Peptid übertragen, und in der S -Stelle befindet sich sie in ARNT „leer“, bevor sie nach der Lieferung der Aminosäure an den Cytosol freigesetzt wird.
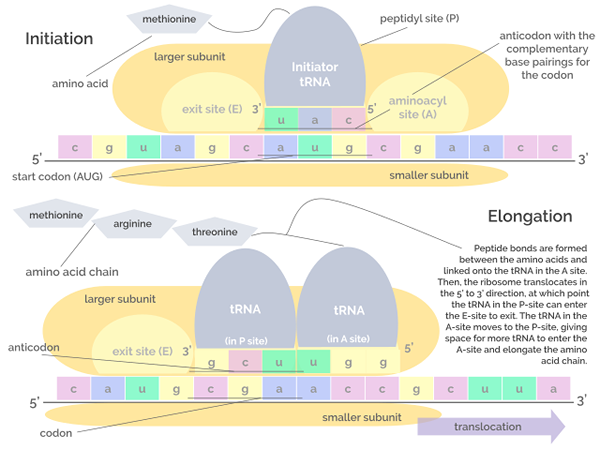 Grafische Darstellung der Initiations- und Dehnungsphasen der Übersetzung (Quelle: Jordan Nguyen/CC BY-S (https: // createRecommons.Org/lizenzen/by-sa/4.0) über Wikimedia Commons)
Grafische Darstellung der Initiations- und Dehnungsphasen der Übersetzung (Quelle: Jordan Nguyen/CC BY-S (https: // createRecommons.Org/lizenzen/by-sa/4.0) über Wikimedia Commons) Verlängerung
Diese Phase besteht aus der "Bewegung" des Ribosoms entlang des mRNA.
Dieser Prozess erfordert einen Faktor.
Kann Ihnen dienen: Okazaki -FragmenteDie Transferase -Peptidil -Aktivität von ribosomalen RNAs ermöglicht die Bildung von Peptidbindungen zwischen aufeinanderfolgenden Aminosäuren, die zur Kette zugesetzt werden.
Beendigung
Die Übersetzung endet, wenn das Ribosom einen der Terminierungscodons trifft, da die ARNs diese Codons nicht erkennen (sie codieren keine Aminosäuren). Es werden auch Proteine, die als Freisetzungsfaktoren bekannt sind.
Prozessübersetzung (Passerprozesse)
In Prokaryoten werden wie in eukaryotischen Zellen die Ribosomen, die für die Proteinsynthese zuständig sind Encodifys es nimmt zu.
Obwohl es in diesen Organismen kein äußerst häufiger Prozess ist, kann die während der Transkription erzeugte primäre RNM nach der registrierenden Reifung nach "Schneiden und Spleißen" leiden. Am häufigsten besteht jedoch darin, Ribosomen zu beobachten, die mit dem primären Transkribieren verbunden sind, das sie gleichzeitig mit der entsprechenden DNA -Sequenz transkribiert wird.
In Anbetracht des oben genannten Beginns beginnt die Übersetzung in vielen Prokaryoten mit dem Ende 5 ', da das 3' Ende der mRNA mit der Form -DNA verbunden bleibt (und gleichzeitig mit der Transkription auftritt).
Nicht übertriebene Regionen
Prokaryotische Zellen produzieren auch RNM mit nicht translatierten Regionen, die als "Shine-Dalgarno-Box" bezeichnet werden und deren Konsensequenz Aggagg ist. Wie ersichtlich ist, sind die UTR -Regionen von Bakterien erheblich kürzer als die von eukaryotischen Zellen, obwohl sie ähnliche Funktionen während der Translation ausüben.
Verfahren
In Bakterien und anderen prokaryotischen Organismen ähnelt der Translationsprozess dem von eukaryotischen Zellen ziemlich. Es besteht auch aus drei Phasen: Initiierung, Dehnung und Beendigung, die von spezifischen prokaryotischen Faktoren abhängen, die sich von denen von Eukaryoten unterscheiden.
Die Verlängerung hängt beispielsweise von Dehnungsfaktoren ab, die als EF-TU und EF-TS bekannt sind, anstelle des eukaryotischen Faktors G.
Verweise
- Alberts, geb., Johnson, a., Lewis, J., Raff, m., Roberts, k., & Walter, p. (2007). Biologie des Zellmolekulares. Garlandwissenschaft. New York, 1392.
- Ton, s. & Braun, w. (2008) Translation: DNA zu mRNA zu Protein. Nature Education 1 (1): 101.
- Griffiths, a. J., Wessler, s. R., Lewontin, r. C., Gelbart, w. M., Suzuki, d. T., & Miller, J. H. (2005). Eine Einführung in die genetische Analyse. Macmillan.
- Lodisch, h., Berk, a., Kaiser, c. ZU., Krieger, m., Scott, m. P., Bretscher, a.,… & Matsudaira, p. (2008). Molekulare Zellbiologie. Macmillan.
- Nelson, d. L., Lehninger, a. L., & Cox, m. M. (2008). Lehninger Prinzipien der Biochemie. Macmillan.
- Rosenberg, l. UND., & Rosenberg, D. D. (2012). Menschliche Gene und Genome: Wissenschaft. Gesundheit, Gesellschaft, 317-338.
- « Sorbus Aria -Eigenschaften, Lebensraum, Eigenschaften, Kultur
- Boletus Aereus Merkmale, Lebensraum, Identifikation, Rezepte »

