Uracil -Struktur, Funktionen, Eigenschaften, Synthese
- 3150
- 587
- Medine Kedzierski
Er Uracil Es ist eine Pyrimidin -Stickstoffbase, die in Ribonukleinsäure (RNA) vorkommt. Dies ist eine der Eigenschaften, die die RNA von Desoxyribonukleinsäure (DNA) unterscheiden, da letztere Timina anstelle von Uracil hat. Sowohl Substanzen, Uracil als auch Timina, unterscheiden sich nur, dass die zweite eine Methylgruppe hat.
Aus evolutionärer Sicht wurde vorgeschlagen, dass RNA das erste Molekül war, das genetische Informationen speicherte und als Katalysator in den Zellen vor DNA und Enzymen arbeitete. Aus diesem Grund wird angenommen, dass Uracil eine Schlüsselrolle in der Entwicklung des Lebens hatte.
 Quelle: Kemikungen [Public Domain]
Quelle: Kemikungen [Public Domain] In Lebewesen wird das Uracil nicht frei gefunden, sondern häufig Monophosphat -Nukleotide (UMP), Diphosphat (UDP) und Trphosphat (UTP). Diese Uracil -Nukleotide haben unterschiedliche Funktionen wie die RNA.
[TOC]
Struktur und Eigenschaften
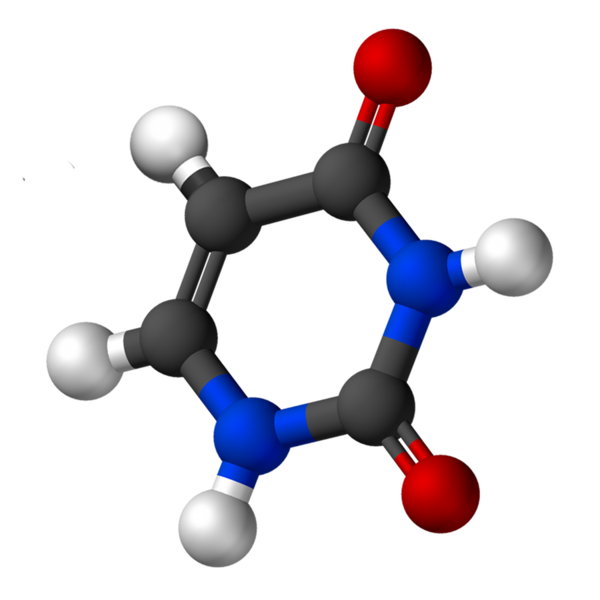
Uracil, als 2,4-Dioxipiridin bezeichnet, hat die empirische Formel C4H4N2ENTWEDER2, dessen Molekulargewicht 112,09 g/mol beträgt und als weißes Pulver gereinigt wird.
Die Struktur von Uridin ist ein heterocyclischer Ring mit vier Kohlenstoffatomen und zwei Stickstoff mit doppelten alternativen Bindungen. Es ist planar.
Es hat eine 50 mg/ml -Löslichkeit, 25 ºC, in 1 m Natriumhydroxid und eine PKA zwischen 7.9 und 8.2. Die Wellenlänge, bei der ihre maximale Absorption auftritt (ʎMax) Es ist zwischen 258 und 260 nm.
Biosynthese
Es gibt einen gemeinsamen Weg für die Biosynthese von Pyrimidin -Nukleotiden (Uracil und Cytokin). Der erste Schritt ist die Biosynthese von Carbamoil -Phosphat von CO2 und NH4+, das durch Carbamoil -Synthetasephosphat katalysiert wird.
Pyrimidin wird aus Carboil -Phosphat und Aspartat gebaut. Beide Substanzen reagieren und bilden N-Carbamoilaspartato, die durch das Transcabamoilasa-Aspartat (ATCASA) katalysierte Reaktion. Der Verschluss des Pyrimidinrings kann durch die Dihydrootase dehydratisiert werden und erzeugt L-Dihydrotatoat.
Das L-Dihydrotoat wird oxidiert und in Orotat verwandelt; Der Elektronenakzeptor ist der NAD+. Es ist eine Reaktion, die durch Dihydroorotat -Dehydrogenase katalysiert wird. Der nächste Schritt besteht in der Übertragung der Fosforibosylgruppe, von Phosphoribosylpyrophosphat (PRPP) bis zu Orotat. Gradidilato -Form (WPO) und anorganische Piroffosphat (PPI), die durch die Fosforibosylorrotat -Transferase katalysiert wurden.
Der letzte Schritt besteht aus der Decarboxylierung des Pyrimidinringes der Gruppe (WPO). Uridilaato-Form (Uridin-5'-Monophosphat, Ump), das durch eine Decarboxylase katalysiert wird.
Dann wird durch die Teilnahme einer Kinase eine Phosphatgruppe vom ATP auf den UMP übertragen und bildet UDP (Uridin-5'-Diphosphat). Letzteres wird wiederholt und bildet UTP (Uridin-5'-Triffosphat).
Kann Ihnen dienen: Zweige der Biologie und was studieren sieBiosyntheseregulation
In Bakterien erfolgt die Regulation der Pyrimidin -Biosynthese durch negatives Feedback auf Transcabamoilasa -Aspartatebene (ATCASA) (ATCASA).
Dieses Enzym wird durch CTP (Citidin-5'-Trophosphat) gehemmt, dem Endprodukt des Biosynthesewegs von Pyrimidinen. ATCAs.
Bei Tieren erfolgt die Regulation der Pyrimidinbiosynthese durch negatives Rückkopplung auf der Ebene von zwei Enzymen: 1) Carbamoil -Synthase -II -Phosphat, das durch UTP gehemmt und durch ATP und PRPP aktiviert wird; und 2) die OMP -Discarboxylase, die durch das Produkt der Reaktion, die sie selbst katalysiert, gehemmt wird, der Ump. Die Biosynthesegeschwindigkeit des OMP hängt von der Verfügbarkeit von PRPP ab.
Funktion in der Biosynthese von RNA
Uracil ist in allen Arten von RNA vorhanden, wie z. Die Biosynthese dieser Moleküle erfolgt durch einen Prozess, der als Transkription bezeichnet wird.
Während des Transkripts werden die in der DNA enthaltenen Informationen in der RNA über eine PON -Polymerase -RNA kopiert. Der umgekehrte Prozess, bei dem die in der RNA enthaltenen Informationen in der DNA kopiert werden.
RNA -Biosynthese benötigt Tryposphat -Nucleoside (NTP), nämlich: Tryphosphat -Uridin (UTP), Tryposphat -Cytidin (CTP), Adenin -Tryposphat -Adenin (ATP) und Tryphosphat -Guanin (GTP). Die Reaktion ist:
(RNA)n Abfall + NTP -> (RNA)N+1 Abfall + PPI
Die Hydrolyse der anorganischen Pyrophosphat (PPI) liefert Energie für die Biosynthese der RNA.
Funktion in der Zuckerbiosynthese
Zuckerester sind in lebenden Organismen sehr häufig. Einige dieser Ester sind die Diphosphas von Nucleosidester wie UDP-Sugar, die in Zellen sehr häufig vorkommen. UDP-Zucker beinhaltet die Biosynthese von Disacchariden, Oligosacchariden und Polysacchariden.
In Pflanzen erfolgt die Biosynthese von Saccharose auf zwei Arten: ein Haupt- und eine sekundäre.
Die Hauptroute besteht in der Übertragung des D-Glucoses von der UDP-D-Glucosa auf die D-FRUCEASE, um Saccharose und UDP zu bilden. Die sekundäre Straße umfasst zwei Stufen: Sie beginnt mit UDP-D-Glucose und Fructose-6-Phosphat und gipfelt mit der Bildung von Saccharose und Phosphat.
In den Brustdrüsen erfolgt die Lactose-Biosynthese aus UDP-D-Galactose und Glukose.
Kann Ihnen dienen: Zweige der BiochemieIn Pflanzen wird die Cellulose-Biosynthese durch kontinuierliche Kondensation von Beta-D-Glycosylabfällen durchgeführt, von UDP-Glucose bis zum nicht reduzierenden Ende der wachsenden Polyglucosekette. In ähnlicher Weise erfordert die Amylose- und Amylopektin-Biosynthese UDP-Glucose als Glucose-Donor-Substrat für die wachsende Kette.
Bei Tieren werden sowohl UDP-Glucose als auch ADP-Glucose für die Glykogenbiosynthese verwendet. In ähnlicher Weise erfordert die Chondroitin-Sulfat-Biosynthese UDP-Xylose, UDP-Galactose und UDP-Glucuronato.
Funktion bei der isomeren Interkonversion von Zucker
Die Umwandlung von Galactose in einen Vermittler der Glykolyse erfolgt über die Leloir -Straße. Einer der Schritte dieser Route ist durch das UDP-Galactose-4-Epimerase-Enzym katalysiert, das die Interkonversion von UDP-Galactose zu UDP-Glucose erleichtert.
Funktion bei der Biosynthese von Glykoproteinen
Während der Biosynthese von Glykoproteinen gehen Proteine durch die CIS-, Medium- und Transsäcke des Golgi -Apparats.
Jeder dieser Taschen hat eine Reihe von Enzymen, die Glykoproteine verarbeiten. Zuckermonomere wie Glukose und Galactose.
Die Nukleotide-Hexose werden durch Antiporte in Golgi-Panzer transportiert. Die UDP-Galactose (UDP-Gal) und die UDP-N-Acetylgalactosamina (UDP-Galnac) betreten durch den Austausch durch Umt die Panzer aus dem Cytosol.
Im Golgi -Tank hydrolysiert eine Phosphatase eine Phosphatgruppe der UDP- und UMP- und PI -Form. Das UDP stammt aus den von Galactosyltransferase und N-Acetylgalactosamiltransferase katalysierten Reaktionen. Die von Phosphatase gebildete UMP dient dazu, Nukleotide-Hexose auszutauschen.
Funktion bei der Regulierung der Synthase
Ein Mechanismus zur Regulierung der Glutamin -Synthase ist die kovalente Modifikation, die aus Adenilation besteht, die inaktiv und flankieren, die es aktiv. Diese kovalente Modifikation ist reversibel und durch Adenyltransferase katalysiert.
Die Aktivität der Adenyltransferase wird durch die Vereinigung des PII -Proteins moduliert, das durch eine kovalente Modifikation Uridinilation reguliert wird.
Sowohl Uridililation als auch der Versatz werden von der Uridililtransferase durchgeführt. In diesem Enzym ist die Uridrationsaktivität auf Glutamin und Phosphat zurückzuführen und wird durch die Vereinigung von Alpha-Zotoglutarat und ATP an die PII aktiviert.
Funktion in der RNA -Ausgabe
Einige RNM werden vor der Übersetzung bearbeitet. In einigen eukaryotischen Organismen, wie z Trypanosoma Brucei, Es gibt Ausgabe der RNA des Transkripts des Untereinheit II -Gens der Cytochromoxidase. Dies geschieht durch Einsetzen von Uracil -Abfällen, eine Reaktion, die durch die terminale Uridransferase katalysiert wurde.
Kann Ihnen dienen: Locken: Komposition, Teile, WichtigkeitEine Leitfaden -RNA, ergänzt zum bearbeiteten Produkt, wirkt für den Bearbeitungsprozess als gemäßigt. Die zwischen dem anfänglichen Transkript gebildeten Basispaare impliziert Basispaare g = u, die nicht Watson-Rekord sind und in der RNA häufig sind.
UDP-Glucose-Biosynthese
Unter physiologischen Bedingungen ist die Glykogenbiosynthese aus Glucose-1-phosphat thermodynamisch unmöglich (positiv ΔG). Aus diesem Grund tritt vor der Biosynthese die Aktivierung von Glucose-1-phosphat (G1P) auf. Dieses G1P und UTP kombiniert die Reaktion zur Bildung von Glucose-Diphosphat-Uridin (UDP-Glucose oder UDPG).
Die Reaktion wird durch die Pyrophosphorlase des UDP-Glucoses katalysiert und ist wie folgt:
G1P + UTP -> UDP -glucosa + 2PI.
Gibbs freie Energievariation in diesem Schritt ist groß und negativ (-33,5 kJ/mol). Während der Sauerstoffreaktion greift das G1P das Alforo Alpha-Phosphor-Atom des UDP-Glucoses und des anorganischen Pyrophosphat (PPI) an. Dann wird der PPI durch eine anorganische Pyrophosphate hydrolysiert, deren Hydrolyseenergie diejenige ist, die die allgemeine Reaktion antreibt.
UDP-Glucose ist eine "hohe Energie" -Substanz. Es ermöglicht die Bildung der glykosidischen Bindungen zwischen dem Glukoserest und der zunehmenden Polysaccharidkette. Das gleiche Energieprinzip gilt für die Reaktionen, an denen die UDP-Zucker teilnimmt, wie die Biosynthese von Disacchariden, Oligosacchariden und Glykoproteinen.
Uracil -DNA -Glycosilasa
Es gibt DNA -Läsionen, die spontan auftreten. Eine dieser Läsionen ist der Sprontane. In diesem Fall erfolgt die Reparatur aufgrund der modifizierten DNA -Base mittels eines Enzyms namens Uracil -DNA -Glycosilasa.
Das Enzym Uracil DNA Glycosilasa eliminiert das beschädigte Zytokin (Uracil) und erzeugt einen Desoxyriboserest, der die Stickstoffbasis fehlt, die als AP-Stelle bezeichnet wird (apurinisch-apirimidinische Stelle).
Dann macht das Endonuklease-AP-Enzym im Phosphodiester-Skelett der AP-Stelle einen Schnitt, wodurch der Zucker-Phosphat-Rest eliminiert wird. DNA -Polymerase I stellt den beschädigten Strang wieder her.
Verweise
- Bohinski, r. 1991. Biochemie. Addison-Wesley Iberoamericana, Wilmington, Delaware.
- Devlin, t.M. 2000. Biochemie. Redaktionsreverté, Barcelona.
- Lodisch, h., Berk, a., Zipurski, s.L., Matsudaria, p., Baltimore, d., Darnell, J. 2003. Zell- und Molekularbiologie. Editorial Medica Panamericana, Buenos Aires, Bogotá, Caracas, Madrid, Mexiko, Sāo Paulo.
- Nelson, d. L., Cox, m. M. 2008. Lehninger-Prinzipien der Biochemie. W.H. Freeman, New York.
- Voet, d. und voet, j. 2004. Biochemie. John Wiley und Söhne, USA.

